- 논문 제목: U-Net: Convolutional Networks for Biomedical Image Segmentation (U-Net: 생물의학 이미지 분할을 위한 컨볼루션 신경망)
- 저자: Olaf Ronneberger, Philipp Fischer, Thomas Brox
- 게재된 학술지/학회: Medical Image Computing and Computer-Assisted Intervention (MICCAI)
- 발표 연도: 2015년
- 논문 링크: https://arxiv.org/pdf/1505.04597
Summary
U-Net 은 의료 데이터에 대한 Segmentation 문제를 해결하는 모델이다.
U-Net 이전까지의 SOTA는 단순히 슬라이딩 윈도우 방식으로 한픽셀씩 움직이면서 분류 문제를 풀고, 컨볼루션의 가운데에 있던 픽셀을 분류문제의 정답으로 하는 방법을 가지고 있었다. 이는 속도를 굉장히 느리게 만드는 문제가 있었고, 전체 콘텍스트를 잘 잡지 못하는 문제가 있었다. 이를 개선한 FCN은 인코더와 디코더 방식으로 되어있지만, 비대칭적인 인코더와 디코더를 사용하고, 스킵커넥션을 덧셈으로 연결해 정보 손실이 U-Net 보다 컸다.
U-Net은 대칭적인 인코더와 디코더의 구조와 Concatenation을 이용한 Skip connection으로 새로운 SOTA를 달성하고 심지어 빠른 속도를 자랑한다.
Network Architecture
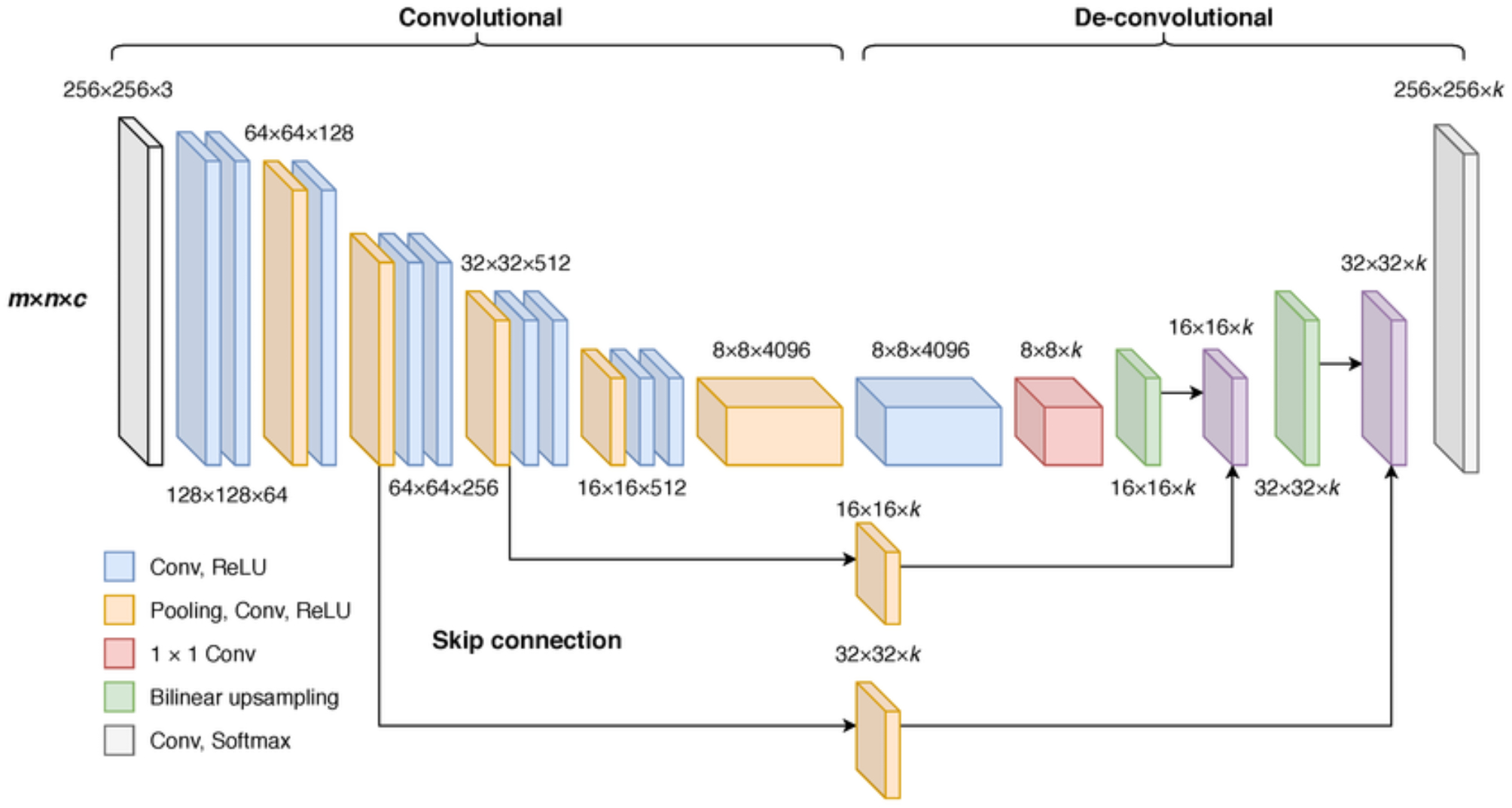
U-Net은 기본적으로 FCN을 기반으로 해서 만들어졌는데, 위에 있는것이 FCN의 네트워크 구조이다.

사실 이 그림 하나만으로 U-Net의 모든 아키텍쳐 설명은 끝난다고 볼 수 있다.
U-Net의 3x3 컨볼루션과 Conv과 pool이 반복적으로 이어지면서 채널 수가 두배가 되고, 사이즈가 두배가 작아지는 인코더 부분은 사실상 VGGNet의 축소 버전이라고 할 수 있다.
U-Net의 가장 핵심적인 부분은 여기에 있는데, VGG와 비슷하게 축소한 압축된 이미지에서 up-conv 2x2를 이용해 이미지를 가로세로 각각 2배 크게 만들고, 채널 수를 반토막으로 줄인다. conv 3x3, ReLU를 적용하는 구조를 반복해, 대칭적으로 다시 원본 크기를 복구한다. 최종적으로 예측하고 싶은 종류인 K개의 채널로 conv 1x1을 이용하여 변환한다.
FCN과 U-Net의 핵심적으로 다른 부분은 다음과 같다.
- FCN은 Skip conneciton을 덧셈을 통해 하지만, U-Net은 채널축으로 Concatenation이 일어난다.
- FCN은 인코더가 깊고 디코더가 얕은 비대칭인 반면, U-Net은 인코더와 디코더가 대칭을 이룬다.
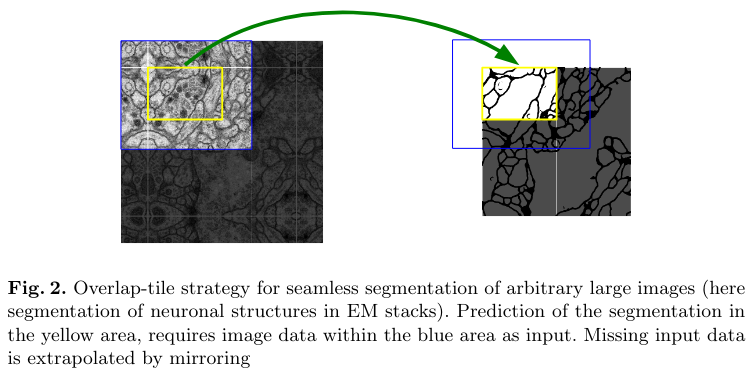 U-Net은 padding이 적용되지 않은 conv layer를 사용하여, 연산을 하면 할수록 출력 이미지의 크기는 점점 작아지게 되는데, 논문에서는 이를 이용하여 주변 콘텍스트를 충분히 받게 하여 더 작은 출력부분만 에측한다. 이를 통해 큰 이미지를 주변 맥락을 얻으면서 여러개로 나눠 추론하여 큰 이미지에 대한 추론을 얻을 수 있다.
U-Net은 padding이 적용되지 않은 conv layer를 사용하여, 연산을 하면 할수록 출력 이미지의 크기는 점점 작아지게 되는데, 논문에서는 이를 이용하여 주변 콘텍스트를 충분히 받게 하여 더 작은 출력부분만 에측한다. 이를 통해 큰 이미지를 주변 맥락을 얻으면서 여러개로 나눠 추론하여 큰 이미지에 대한 추론을 얻을 수 있다.
Training
Caffe 프레임워크에 구현된 SGD로 학습함.
U-Net이 의료 데이터를 위한 모델이므로, 고해상도 이미지를 받기 위해 입력 이미지 타일의 크기를 최대화 하고, GPU의 한계가 있으므로 Batch Size를 1로, 즉 단일 이미지로 줄였다.
Batch size가 1이면 학습이 불안정해 지는데, 이를 막기위해 굉장히 높은 모멘텀인 0.99를 사용하여 과거의 학습 샘플을 많이 참고하여 안정적으로 학습할 수 있게 하였다. with 일때, soft-max 는 다음과 같다. ( 는 픽셀에서 의 k번째 채널의 activation)
Energy Function은 다음과 같다. 즉, Weighted cross entropy 와 같다.
은 각 픽셀의 true label을 돌려주는 함수고, 은 픽셀별로 가중치를 반환하는 함수이다. 가중치는 다음과 같이 정의된다.
가중치는 정답 레이블에서 미리 계산된다. 는 클래스간 빈도 불균형을 해결하기 위한 weight map이다. 은 가장 가까이 있는 셀과의 거리이고, 는 두번째로 가까이 있는 셀과의 거리이다. 이 두개의 함수를 이용하여 모델은 두 세포 사이에 있는 경계에 더 큰 가중치를 부여 하게 된다. 논문에서의 experiments에서는 and 로 설정했다.

더 큰 가중치가 부여된 모습.
가중치 초기화는 널리 알려진 SD를 (where N denotes the number of incoming nodes of one neuron)으로 하는 정규분포로 한다.
Data Augmentation
Shift, Rotation, Gray value variations, Drop-out(암시적 데이터 증강으로 봄), Elastic Deformation 등을 활용한다. 논문에서는 특히 Elastic Deformation이 적은 량의 데이터셋으로도 학습을 시키는 주요 기법이라고 한다.

이것이 Elastic deformation의 예시 사진인데, U-Net 논문에서는 안보여줘서 따로 찾아왔다. 확실히 세포같은 데이터에 적용하면 잘 작동할 증강기법인듯…
Experiments
의학 데이터 용어가 너무 많아서 이해를 잘 못했다…
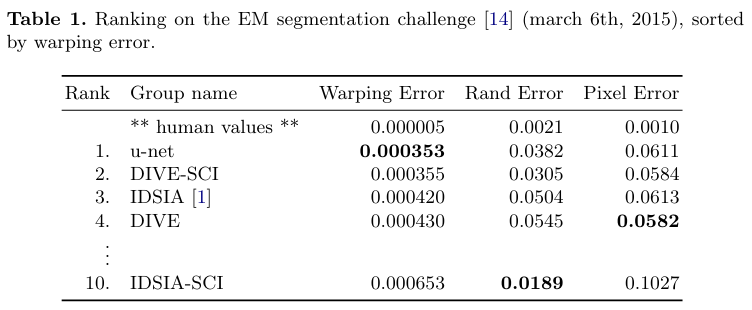
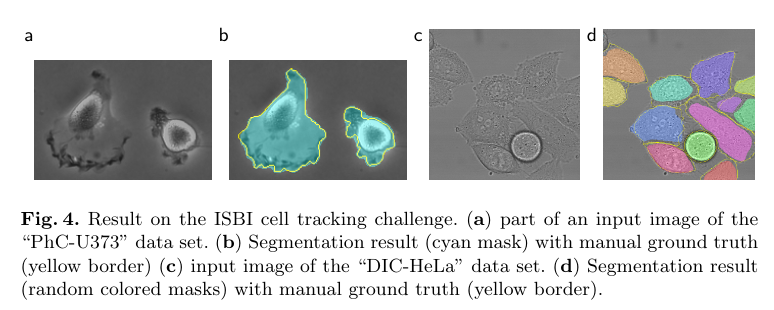
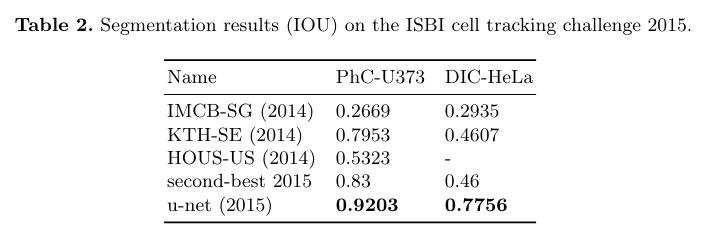
Conclusion
U-Net은 생물의학 이미지 분할을 위해 설계된 강력한 완전 컨볼루션 신경망 아키텍처를 제안했다.
이 논문의 가장 큰 의의는 대칭적인 인코더-디코더 구조와 skip connection을 결합하여, 매우 적은 양의 학습 데이터만으로도 정교한 분할 성능을 달성할 수 있음을 입증한 데에 있다. 특히 데이터가 부족한 의료 영상 분야에서 딥러닝의 실질적인 적용 가능성을 크게 확장시킨 중요한 연구라 할 수 있다.
U-Net은 그 독창적인 구조와 뛰어난 성능으로 인해 발표 이후 수많은 후속 연구의 기반이 되었으며, 현재까지도 의료 영상 분할 분야의 사실 표준으로 자리매김하고 있는 기념비적인 논문이다.